استفاده از روش های بیوانفورماتیکی یا روش های کامپیوتری باعث میشود که ما بتوانیم آزمایش عملی خود را با بهترین و بهینه ترین حالت طراحی کنیم. SNP ها هم از این مورد مثتثنی نیستند. روش های مختلفی برای آنالیز کامپیوتری SNP ها وجود دارد که در این نوشته با ابزارهای مختلف آنالیز In silico برای SNP ها آشنا خواهیم شد :
اسنیپ ها :روش ها و اهمیت مطالعه
رایج ترین تغییرات ژنتیکی در ژنوم که میتوانند بروز تفاوت ها درخصوصیات فردی و استعداد ابتلا به بیماری ها را تحت تاثیر قرار دهند چند شکلیهای تک نوکلئوتیدی (اسنیپ ها) هستند. چندشکلیها به طور طبیعی در DNA ژنومی انسان وجود دارند و در شناسایی تفاوتهای فردی در ارتباط با استعداد ابتلا به بیماریها مورد استفاده قرار میگیرند. وجود این چندشکلیها در ژن های مختلف می تواند منجر به بروز بیماری میگردد. بنابراین وجود این چند شکلی در ژنهای مذکور میتواند خطر ابتلا به بیماری ها و سایر خصوصیات فردی افراد را تحت تاثیر قرار دهد. مطالعه و تعیین وضعیت اسنیپ ها با استفاده از تکنیک های مختلف مولکولی نظیر PCR-RFLP، Tetra ARMs-PCR، توالی یابی، HRM-real time PCR و Real time PCR-Probe در آزمایشگاه صورت می گیرد. تعیین وضعیت اسنیپ ها با استفاده از روش های آزمایشگاهی مذکور در مطالعات مختلف نظیر موردی-شاهدی و مطالعه بیماران می تواند منجر به کشف واریانت های مرتبط با بیماری ها و خصوصیات فردی افراد گردد. بررسی چگونگی اعمال تاثیر وجود یک اسنیپ در یک ژن بر روی عملکرد و یا بیان آن ژن می تواند منجر به شناخت و درک عمیق ما در ارتباط با اثرات وجود اسنیپ ها در نواحی مختلف ژنومی گردد. در اینجا ما با ارایه یک فلوچارت مناسب به چگونگی بررسی و آنالیز In Silico اسنیپ ها می پردازیم. مطالعه آزمایشگاهی و بیوانفورماتیکی (In silico) اسنیپ ها می تواند زمینه را برای کشف مارکرهای ژنتیکی مرتبط با بیماری ها (پزشکی شخص محور) فراهم نماید.
In Silico Analysis چیست؟
مطالعه اسنیپ ها با استفاده از ابزارهای مختلف بیوانفورماتیکی In Silico Analysis نامیده می شود. این ابزارها اکثرا به صورت آنلاین در خدمت پژوهشگران هستند.
داده کاوی یا جمع آوری اطلاعات
به منظور انالیز بیوانفورماتیکی اسنیپ ها ما بایستی اطلاعات لازم مربوط به توالی DNA و توالی پروتئین های ژن های در بردارنده اسنیپ ها را بدست آوریم. برای دسترسی به اطلاعات SNP های مورد مطالعه، پایگاه داده NCBI SNP (https://www.ncbi.nlm.nih.gov/SNP) به کار گرفته می شود. این دسترسی شامل اطلاعات SNP ها، از جمله فرمت توالیهای DNA و پروتئین ژن های در بردارنده اسنیپ ها در قالب FASTA است.
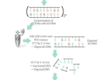
در شکل زیر فلوچارت نرم افزارهای مورد نیاز برای آنالیز In Silico اسنیپ ها را مشاهده می کنید.

برای تجزیه و تحلیل و تحلیل SNP های غیر هم معنی یا کد کننده (nsSNP) از ابزارهای بیوانفورماتیک مختلف استفاده می گردد که شامل SIFT، PolyPhen-2، PhD-SNP، SNPs&GO، I-Mutant، MUpro و Project HOPE می باشند. در طرف مقابل برای انالیز اسنیپ های غیر کد کننده یا هم معنی و همچنین SNP های واقع در مناطق غیر ترجمه شونده (به اختصار UTR) ابزارهای FastSNP و SNP Function Prediction مورد استفاده قرار می گیرند. نمودار فلوچارت نرمافزارهای بیوانفورماتیک مورد استفاده در شکل فوق نشان داده شده است. در اینجا ما با اختصار به توضیح این فلوچارت و ابزارهای بیومانفورماتیکی در فلوچارت ارائه شده می پردازیم.
GeneMANIA
GeneMANIA (http://www.genemania.org/) یک نرمافزار بیوانفورماتیک آنلاین است که عملکرد ژن را پیشبینی میکند و اطلاعاتی را در زمینههای مختلف، برهمکنشهای فیزیکی، برهمکنش های ژنی، دومین های پروتئینی به اشتراک گذاشته شده، وضعیت بیان هم زمان ژن ها نسبت به یکدیگر، موقعیت قرار گیری شان، دامنههای مشترک و مسیرهای درگیردر شبکه ژنی را فراهم میکند. برای بدست آوردن اطلاعات مذکور کافیست تنها اسم ژن مورد نظر را در قسمت گفته شده نرم افزار وارد نموده و نتایج را در قالب PDF دریافت نمایید شما می توانید با اعمال فیلتر اطلاعات شبکه ای ژن مورد نظر با سایر ژن های دیگر را انتخاب و اطلاعات مورد نیاز خود را بدست آورید.
آنالیز In silico اسنیپ های کد کننده یا غیر هم معنی
نرم های های مورد استفاده جهت پاتوژنتیک بودن اسنیپ ها
برای پیش بینی پاتوژنیکی SNP های کد کننده یا بد معنی، از چند ابزار آنلاین استفاده می شود که شامل موارد زیر می باشند:
SIFT
SIFT (http://sift.jcvi.org/) پیش بینی می کند که آیا جایگزینی یک اسید آمینه اثر مخربی بر عملکرد پروتئین دارد یا خیر. SIFT به طور خودکار چندین مرحله را با استفاده از NCBI PSI-BLAST انجام می دهد. SIFT یک امتیاز احتمالی برای اسنیپ مورد مطالعه که منجر به تغییر یک اسید آمینه شده می دهد اگر نمره داده شده کمتر یا مساوی از حد 0.05 باشد نشان دهنده زیان بار بودن وجود اسنیپ مورد می باشد وامتیاز بزرگتر از آن حد قابل تحمل می باشد.
PolyPhen v2
PolyPhen v2 (http://genetics.bwh.harvard.edu/pph 2/) اثر احتمالی SNP های کد کننده یا بد معنی را بر اساس توالی، تغییر ساختار و ویژگیهای فیلوژنتیکی پیشبینی میکند و خروجی پیشبینی PolyPhen v2 احتمال آسیبرسان یا خوشخیم بودن اسنیپ مورد مطالعه به همراه یک امتیاز عددی از 0.0 (خوش خیم) تا 1.0 (مضر) می باشد.
PhD-SNP
PhD-SNP (http://snps.biofold.org/phd-snp/phd-snp.html) مبتنی بر ماشینهای محاسباتی پشتیبان (SVMs) است که پیشبینی میکند آیا یک پلیمورفیسم کد کننده یا بد معنی در قالب یک جهش، طبیعی است یا با اختلالات ژنتیکی در انسان مرتبط می باشد (طبیعی و یا بیماری زا بودن اسنیپ مورد نظر را مشخص می نماید).
SNPs&GO
SNPs & GO (http://snps-and-go.biocomp.unibo.it/snps-andgo/) نیز یک روش دقیق مبتنی بر ماشین محاسباتی پشتیبان (SVM) است که برای پیشبینی اینکه آیا وجود اسنیپ مورد نظر که منجر به جایگزینی اسید آمینه شده است می تواند با بروز بیماریها مرتبط باشد یا خیر، استفاده میشود. امتیاز احتمالی بالاتر از 0.5 نشان می دهد که SNP مرتبط با بروز بیماری است.
تجزیه و تحلیل تاثیر اسنیپ ها بر پایداری پروتئین
تغییر اسید آمینه می تواند باعث تغییر در پایداری ساختاری پروتئین گردد که می تواند بر روی عملکرد پروتئین دخالت داشته باشد. به منظور تجزیه و تحلیل پایداری پروتئین ناشی از اسنیپ های غیر هم معنی یا کد کننده از دو سرور آنلاین استفاده می گردد که شامل I-Mutant 3.0 و MUproمی باشند.
I-mutant 3.0
ابزار I-Mutant 3.0 (http: //gpcr.biocomp.unibo. it/cgi/predictors/I-Mutant3.0/I-Mutant3.0.cgi) برای تعیین تاثیر اسنیپ های کد کننده یا غیر هم معنی بر روی پایداری پروتئین با پیشبینی اثر تغییر اسید آمینه بر مقدار تغییر انرژی آزاد (Delta Delta G: DDG) بر اساس توالی یا ساختار سوم پروتئین استفاده می گردد. توالی FASTA پروتئین، تغییر اسید آمینه و موقعیت جهش به عنوان ورودی برای پیشبینی تأثیر جهش بر پایداری پروتئین استفاده می گردد. I-Mutant 3.0 خروجی را به عنوان مقدار DDG می دهد که در یکی از سه حالت پیش بینی طبقه بندی می شود: تا حد زیادی ناپایدار (DDG <- 0.5 کیلوکالری/ مول)، تا حد زیادی پایدار (DDG > 0.5 کیلوکالری / مول)، یا خنثی (- 0.5 ≤ DDG ≤ 0.5 کیلوکالری / مول).
MUpro
ابزار دوم جهت بررسی و انالیز پایداری MUpro (http:// mupro.proteomics.ics.uci.edu/) می باشد. این سرور مبتنی بر دو روش learning ماشینی است: ماشینهای محاسباتی پشتیبان و شبکههای عصبی. هر دوی آنها بر روی یک مجموعه داده جهش بزرگ تعریف شده اند و دقت بالای 84٪ را نشان می دهند. این سرور امتیازی بین -1 و 1 را به عنوان سطح اطمینان پیش بینی محاسبه می کند. توالی پروتئین در قالب FASTA ، نام جهش، موقعیت جهش، اسید آمینه اصلی و اسید آمینه جایگزین به عنوان موارد ورودی نرم افزار برای پیشبینی تأثیر جهش بر پایداری پروتئین استفاده می شود. نمره اطمینان کمتر از صفر ( < 0 ) نشان می دهد که جهش باعث کاهش پایداری پروتئین می شود، در حالی که نمره اطمینان بالاتر از صفر( > 0) به این معنی است که جهش پایداری پروتئین را افزایش می دهد.
تجزیه و تحلیل بیوفیزیکی و ساختاری
به منظور تجزیه و تحلیل بیوفیزیکی و ساختاری پروتئین ناشی از وجود اسنیپ های غیر هم معنی یا کد کننده ، از (https://www3.cmbi.umcn.nl/hope/)Project HOPE استفاده می گردد.
Project HOPE
Project HOPE تأثیر اسنیپ های غیر هم معنی یا کد کننده بر ساختار پروتئین و عملکرد مربوطه را تجزیه و تحلیل میکند. توالی پروتئین و جهش به عنوان ورودی برای HOPE استفاده می شود.
آنالیز اسنیپ های موجود در نواحی غیر ترجمه شونده
به منظور تجزیه و تحلیل SNP های واقع در ناحیه غیر ترجمه شونده ژنومی SNP function prediction SNPinfo (FuncPred) و FASTSNP استفاده می گردد.
SNP function prediction SNPinfo (FuncPred)
(http://snpinfo.niehs.nih.gov/snpinfo/snpfunc.htm) SNP function prediction SNPinfo ابزاری برای پیشبینی عملکرد SNP است که بررسی میکند آیا SNP میتواند تنظیم رونویسی را با تأثیر بر فعالیت جایگاه های اتصال فاکتور رونویسی (TFBS) یا تغییر الگوی اسپلایسینگ تغییر دهد یا خیر.
FastSNP
ابزار آنلاین FastSNP (http://FASTSNP.ibms.sinica.edu.tw) برای تعیین تأثیر SNP های واقع در مناطق UTR و سایر مناطق غیر کد کننده استفاده می گردد. این سرور پیشبینی که میکند آیا یک SNP غیرکدکننده، جایگاه اتصال فاکتور رونویسی یک ژن را تغییر میدهد یا خیر. این امتیاز بر اساس سطوح ریسک با رتبهبندی 0، 1، 2، 3، 4 یا 5 داده میشود. این امتیازها نشاندهنده سطوح ریسک به ترتیب (از صفر تا 5) بدون ریسک، ریسک بسیار کم، ریسک کم، ریسک متوسط، ریسک زیاد و ریسک بسیار زیاد است.
جمع بندی
تحلیل بیوانفورماتیکی اسنیپها یک ابزار قدرتمند در زمینه تحقیقات ژنتیکی و بیولوژیکی مولکولی است که امکان ارزیابی و پیشبینی اسنیپها برای عملکرد ژنوم را میکند. این روشها به ما امکان میدهند که مخرب یا مفید اسنیپها بر روی ساختار و عملکردها و ژنوم را تجزیه و تحلیل کنند. از آنجایی که در سطح ژنتیکی میتواند به بیماریها یا ویژگیهای فردی مرتبط باشد، این اطلاعات بیوانفورماتیکی میتواند به بررسی مکانیسمهای بیوشیمیایی بیماریها و همچنین تعیین راهبردهای درمانی و اختصاصی یابند بپردازد. این موارد مهم در توسعه داروها و تصمیم گیری های پزشکی درمانی هستند. به کمک این ابزارها، میتوان بهترین تصمیم را برای دادههای ژنتیکی برای ارتقاء سلامت و بهبود کیفیت زندگی فردی و راهکارهای دقیقتر در هوا و درمان بیماریها ارائه کرد.